Технологическое оборудование и технологические трубопроводы сНиП 3.05.05-84
СТРОИТЕЛЬНЫЕ НОРМЫ И ПРАВИЛА
ГОСУДАРСТВЕННЫЙ КОМИТЕТ СССР
ПО ДЕЛАМ СТРОИТЕЛЬСТВА
РАЗРАБОТАНЫ ВНИИмонтажспецстроем Минмонтажспецстроя СССР (инж. В. Я. Эйдельман, д-р техн. наук В. В. Поповский — руководители темы; кандидаты техн. наук В. И. Оботуров, Ю. В. Попов, Р. И. Тавастшерна), Гипронефтеспецмонтажом Минмонтажспецстроя СССР (канд. техн. наук И. С. Гольденберг) и Гипрохиммонтажом Минмонтажспецстроя СССР (инженеры И. П. Петрухин, М. Л. Эльяш).
ВНЕСЕНЫ Минмонтажспецстроем СССР.
ПОДГОТОВЛЕНЫ К УТВЕРЖДЕНИЮ Отделом технического нормирования и стандартизации Госстроя СССР (инж. Б. А. Соколов) .
С введением в действие СНиП
3.05.05-84 „Технологическое оборудование
и технологические трубопроводы”
утрачивает силу СНиП III. 31.78*
„Технологическое оборудование. Основные положения”.
Основные положения”.
Государственный комитет | Строительные нормы и правила | СНиП 3.05.05-84 |
по делам строительства (Госстрой СССР) | Технологическое оборудование и технологические трубопроводы | Взамен СНиП III-31-78* |
Настоящие правила распространяются на
производство и приемку работ по монтажу
технологического оборудования и
технологических трубопроводов (в
дальнейшем — „оборудование”
и „трубопроводы”),
предназначенных для получения, переработки
и транспортирования исходных, промежуточных
и конечных продуктов при абсолютном
давлении от 0,001 МПа (0,01
кгс/см 2) до 100 МПа
вкл. (1000 кгс/см2), а
также трубопроводов для подачи
теплоносителей, смазки и других веществ,
необходимых для работы оборудования.
(1000 кгс/см2), а
также трубопроводов для подачи
теплоносителей, смазки и других веществ,
необходимых для работы оборудования.
Правила должны соблюдаться всеми организациями и предприятиями, участвующими в проектировании и строительстве новых, расширении, реконструкции и техническом перевооружении действующих предприятий.
Работы по монтажу оборудования и трубопроводов, подконтрольных Госгортсхнадзору СССР, в том числе сворка и контроль качества сварных соединений, должны производиться согласно правилам и нормам Госгортехнадзора СССР.
1.1. При производстве работ по монтажу оборудования и трубопроводов необходимо соблюдать требования СНиП по организации строительного производства, СНиП III-4-80, стандартов, технических условий и ведомственных нормативных документов, утвержденных в соответствии со СНиП 1.01.01-82*.
1.2. Работы по монтажу
оборудования и трубопроводов должны
производиться в соответствии с
утвержденной проектно-сметной и рабочей
документацией, проектом производства
работ (ППР) и документацией
предприятий-изготовителей.
Внесены Минмонтажспецстроем СССР | Утверждены постановлением Государственного комитета СССР по делам строительства от 7 мая 1984 г. № 72 | Срок введения в действие 1 января 1985 г. |
1.3. Монтаж оборудования и трубопроводов должен осуществляться на основе узлового метода строительства и комплектно-блочного метода монтажа.
Примечания: 1. Под узловым методом строительства
понимается организация строительно-монтажных
работ с разделением пускового комплекса
на взаимоувязанные между собой
технологические узлы —
конструктивно и технологически
обособленные части объекта строительства,
техническая готовность которых после
завершения строительно-монтажных работ
позволяет автономно, независимо от
готовности объекта в целом, производить
пусконаладочные работы, индивидуальные
испытании и комплексное опробование
агрегатов, механизмов и устройств.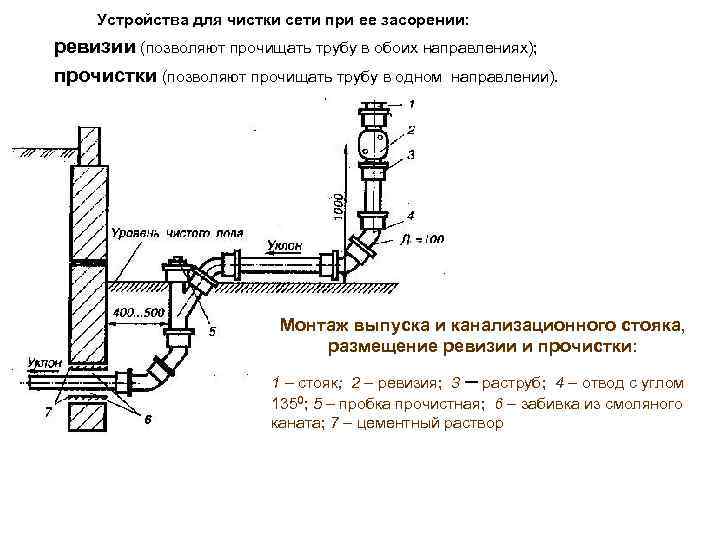
2. Под комплектно-блочным методом монтажа понимается организация монтажа оборудования и трубопроводов с максимальным переносом работ со строительной площадки в условия промышленного производства с агрегированием оборудования, трубопроводов и конструкций в блоки на предприятиях-поставщиках, а также на сборочно-комплектовочных предприятиях строительной индустрии и строительно-монтажных организаций с поставкой на стройки в виде комплектов блочных устройств.
1.4. В документации, выдаваемой в соответствии с п. 1.2 монтажной организации, должны быть предусмотрены:
а) применение технологических блоков и блоков коммуникаций с агрегированием, их составных частей на основе номенклатуры и технических требований, утвержденных или взаимно согласованных вышестоящими организациями заказчика и подрядчика, осуществляющего монтажные работы;
б) разделение объекта строительства на технологические узлы, состав и границы которых определяет проектная организация по согласованию с заказчиком и подрядчиком, осуществляющим монтажный работы;
в) возможность подачи технологических блоков и блоков коммуникаций к месту монтажа в собранном виде с созданием в необходимых случаях монтажных проемов в стенах и перекрытиях зданий и шарнирных устройств в опорных строительных конструкциях для монтажа методом поворота, а также с усилением при необходимости строительных конструкций для восприятия ими дополнительных временных нагрузок, возникающих в процессе монтажа; постоянные или временные дороги для перемещения тяжеловесного и крупногабаритного оборудования, а также кранов большой грузоподъемности;
г) данные по допускам для расчета точности
выполнения геодезических разбивочных
работ и создания внутренней геодезической
разбивочной основы для монтажа
оборудования.
1.5. Генподрядчик должен привлекать монтажную организацию к рассмотрению и составлению заключения по проекту организации строительства, конструктивным решениям зданий и сооружений, а также технологическим компоновкам, в которых должны быть определены возможность и основные условия производства работ комплектно-блочным и узловым методами.
1.6. Генподрядчик должен обеспечить, а монтажная организация — поручить от генподрядчика (или, по согласованию с ним, непосредственно от заказчика) необходимый комплект рабочей документации с отметкой заказчика на каждом чертеже (экземпляре) о принятии к производству.
1.7. Поставку оборудования,
трубопроводов и необходимых для монтажа
комплектующих изделий и материалов
следует осуществлять по графику,
согласованному с монтажной организацией,
где должна предусматриваться первоочередная
поставка машин, аппаратов, арматуры,
конструкций, изделий и материалов,
включенных в спецификации на блоки,
подлежащие изготовлению монтажными
организациями.
1.8. Окончанием работ по монтажу оборудования и трубопроводов надлежит считать завершение индивидуальных испытаний, выполненных в соответствии с разд. 5 настоящих правил, и подписание рабочей комиссией акта приемки оборудования.
После окончания монтажной организацией работ по монтажу, т. е. завершения индивидуальных испытаний и приемки оборудования под комплексное опробование, заказчик производит комплексное опробование оборудования в соответствии с обязательным приложением 1.
1.9. На каждом объекте строительства в процессе монтажа оборудования и трубопроводов следует вести общий и специальные журналы производства работ согласно СНиП по организации строительного производства и оформлять производственную документацию, виды и содержание которой должны соответствовать обязательному приложению 2, а ее формы — устанавливаться ведомственными нормативными документами.
Технологическое оборудование и технологические трубопроводы СНиП 3.
 05.05-84
05.05-84Главная
Информация
Каталоги и справочники
15.06.2012
Настоящие правила распространяются на производство и приемку работ по монтажу технологического оборудования и технологических трубопроводов (в дальнейшем – “оборудование” и “трубопроводы”), предназначенных для получения, переработки и транспортирования исходных, промежуточных и конечных продуктов при абсолютном давлении от 0,001 МПа (0,01 кгс/кв.см) до 100 МПа вкл. (1000 кгс/кв.см), а также трубопроводов для подачи теплоносителей, смазки и других веществ, необходимых для работы оборудования.
Правила должны соблюдаться всеми организациями и предприятиями, участвующими в проектировании и строительстве новых, расширении, реконструкции и техническом перевооружении действующих предприятий. Работы по монтажу оборудования и трубопроводов, подконтрольных Госгортехнадзору, в том числе сварка и контроль качества сварных соединений, должны производиться согласно правилам и нормам Госгортехнадзора.
С введением в действие СНиП 3.05.05-84 „Технологическое оборудование и технологические трубопроводы” утрачивает силу СНиП III. 31.78* „Технологическое оборудование. Основные положения”
Содержание книги Технологическое оборудование и технологические трубопроводы СНиП 3.05.05-84 |
|
Общие положения Подготовка к производству монтажных работ Общие требования Передача в монтаж оборудования, изделий и материалов Приемка под монтаж зданий, сооружений и фундаментов Изготовление сборочных единиц трубопроводов Сборка технологических блоков и блоков коммуникаций Производство монтажных работ Общие требования Монтаж оборудования Монтаж трубопроводов Сварные и другие неразъемные соединения трубопроводов Общие требования Контроль качества соединений стальных трубопроводов Контроль качества соединений трубопроводов из цветных металлов и сплавов Контроль качества соединений пластмассовых трубопроводов Индивидуальные испытания смонтированного оборудования и трубопроводов Приложение 1 Обязательное Порядок производства пусконаладочных работ Приложение 2 Обязательное Производственная документация, оформляемая при монтаже оборудования и трубопроводов Приложение 3 Обязательное Механические испытания сварных образцов стальных трубопроводов Приложение 4 Обязательное Определение суммарного балла качества сварного соединения стального трубопровода по результатам радиографического контроля |
Поделиться ссылкой:
Вернуться к списку
Установка — документация CFSAN SNP Pipeline 2.
 2.1
2.1Программный пакет SNP Pipeline состоит из сценариев Python с зависимостями от исполняемых программ, запускаемых скриптами.
Шаг 1. Требования к операционной системе
Конвейер SNP работает в среде Linux. Это было протестировано на следующих платформах:
- Красная шляпа
- CentOS
- Убунту
Шаг 2. Зависимости исполняемого программного обеспечения
Перед использованием конвейера SNP необходимо установить следующее программное обеспечение.
| Программное обеспечение | Протестированная версия | Описание |
|---|---|---|
| Галстук-бабочка2 | 2.3.4.1 | Инструмент для выравнивания считываний с длинными эталонными последовательностями |
| СМАЛЬТА | 0.7.6 | Инструмент для выравнивания считываний с длинными эталонными последовательностями |
| SAMtools | 1,8 | Утилиты для управления выравниваниями в формате SAM |
| Пикард | 2. 18.4 18.4 | Набор инструментов для работы с данными последовательности |
| ГАТК | 3.8-1-0 | Инструменты для обнаружения вариантов и генотипирования |
| ВарСкан | 2.3.9 | Инструмент для обнаружения вариантов в данных NGS |
| табикс | 1,8 | Универсальный индексатор для файлов положения генома с разделителями табуляцией |
| bgzip | 1,8 | Часть пакета tabix, bgzip — утилита сжатия блоков |
| BcfTools | 1,8 | Утилиты для вызова вариантов и управления VCF и BCF |
Примечание: приведенные выше версии протестированы и работают вместе. Другие версии также могут работать.
Примечание: вам понадобится Bowtie2 или SMALT. Вам не нужно устанавливать оба.
Однако включенные файлы результатов были сгенерированы с помощью Bowtie2. Ваши результаты могут отличаться
при использовании СМАЛТ.
Примечание. Пикард требуется при удалении повторяющихся чтений и при повторном выравнивании чтений вокруг вставок. Обе эти функции включены по умолчанию, но их можно отключить в файле конфигурации.
Примечание. GATK требуется при повторном выравнивании операций чтения вокруг вставок, что включено по умолчанию. но может быть отключен в файле конфигурации.
Шаг 3. Переменные среды
Определите переменную среды CLASSPATH, чтобы указать расположение jar-файлов Picard, VarScan и GATK. Добавлять следующие строки (или что-то подобное) в ваш файл .bashrc:
экспорт ПУТЬ К КЛАССУ=~/software/varscan.v2.3.9/VarScan.jar:$ПУТЬ К КЛАССУ экспорт ПУТЬ К КЛАССУ=~/software/picard/picard.jar:$ПУТЬ К КЛАССУ экспорт CLASSPATH=~/software/GenomeAnalysisTK-3.8-1-0-gf15c1c3ef/GenomeAnalysisTK.jar:$CLASSPATH
Шаг 4 — Python
Конвейер SNP совместим с версиями Python 2.7, 3.4, 3.5, 3.6 и 3.7. Конвейер не тестировался на других версиях Python.
Вы можете либо собрать из исходного кода, либо установить предварительно скомпилированную версию с помощью менеджера пакетов Linux.
Шаг 5 — Pip
Это может быть трудным шагом установки — действуйте осторожно. Инструмент pip используется для установки пакетов Python. включая snp-pipeline и другие пакеты, используемые snp-pipeline. Некоторые новые версии Python включают pip. Проверьте, не установлен ли уже pip:
$ pip -V
Если pip еще не установлен, выполните следующие действия:
Загрузите get-pip.py с https://pip.pypa.io/en/latest/installing.html#install-pip $ python get-pip.py --user
Примечание: избегайте использования sudo при установке pip. У некоторых пользователей возникли проблемы с установкой и загрузкой пакетов, когда pip устанавливается с помощью sudo.
Шаг 6. Установка пакета SNP Pipeline Python
Существует несколько способов установки SNP Pipeline в зависимости от того, собираетесь ли вы работать с исходным кодом или просто запускаете его.
Метод установки 1 для большинства пользователей
Это рекомендуемый метод установки для новых пользователей.
Если вы хотите запустить программное обеспечение без просмотра или изменения исходного кода, следуйте приведенным ниже инструкциям.
В командной строке:
$ pip install --user snp-pipeline
Или, если у вас установлен virtualenvwrapper:
$ mkvirtualenv snp-pipeline $ pip установить snp-конвейер
Способ установки 2 для разработчиков программного обеспечения
Если вы собираетесь работать с исходным кодом в роли разработчика программного обеспечения, вам следует клонировать репозиторий GitHub, как описано в разделе «Содействие» этой документации.
Модернизация конвейера SNP
Если вы ранее устанавливали с помощью pip, вы можете обновить его до новейшей версии из командной строки:
$ pip install --user --upgrade snp-pipeline
Удаление SNP Pipeline
Если вы устанавливали с помощью pip, вы можете удалить из командной строки:
$ pip uninstall snp-pipeline
Советы
Существует зависимость от пакета psutil python. Пип попытается
установите пакет psutil автоматически при установке snp-pipeline.
Если произойдет сбой с сообщением об отсутствующем Python.h, вам нужно будет
вручную установите пакет python-dev.
В Ubuntu используйте эту команду:
Пип попытается
установите пакет psutil автоматически при установке snp-pipeline.
Если произойдет сбой с сообщением об отсутствующем Python.h, вам нужно будет
вручную установите пакет python-dev.
В Ubuntu используйте эту команду:
$ sudo apt-get установить python-dev
Возможно, вам потребуется обновить среду выполнения Java (JRE) для запуска Picard.
Интегрированный конвейер добычи и использования SNP (ISMU) для данных секвенирования следующего поколения
1. Имельфорт М., Эдвардс Д. (2009) De novo секвенирование геномов растений с использованием технологий второго поколения. Кратко Биоинформ 10: 609–618 10.1093/нагрудник/bbp039 [PubMed] [CrossRef] [Google Scholar]
2. Варшней Р.К., Наяк С.Н., Май Г.Д., Джексон С.А. (2009 г.) Технологии секвенирования следующего поколения и их значение для генетики и селекции сельскохозяйственных культур. Тенденции Биотехнологии 27: 522–530 10.1016/j.tibtech.2009.05.006 [PubMed] [CrossRef] [Google Scholar]
3. Маршалл Д.Дж., Хейворд А., Илз Д., Имельфорт М., Стиллер Дж. и др. (2010) Целевая идентификация геномных регионов с использованием TAGdb. Растительные методы
6: 19–19
10.1186/1746-4811-6-19
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Маршалл Д.Дж., Хейворд А., Илз Д., Имельфорт М., Стиллер Дж. и др. (2010) Целевая идентификация геномных регионов с использованием TAGdb. Растительные методы
6: 19–19
10.1186/1746-4811-6-19
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
4. Майкл Т.П., Джексон С. (2013) Первые 50 геномов растений. Plant Gen 6: doi:10.3835/plantgenome2013.03.0001 [Google Scholar]
5. Дюран С., Эпплби Н., Варди М., Имельфорт М., Эдвардс Д. и др. (2009)Обнаружение полиморфизма одиночных нуклеотидов в ячмене с использованием autoSNPdb. Завод Биотехнолог J 7: 326–333 10.1111/j.1467-7652.2009.00407.x [PubMed] [CrossRef] [Google Scholar]
6. Лу Т., Лу Г., Фан Д., Чжу С., Ли В. и др. (2010) Функциональная аннотация транскриптома риса при разрешении одного нуклеотида с помощью RNA-seq. Геном Res 20: 1238–1249 гг. 10.1101/гр.106120.110 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
7.
Дубей А., Фармер А., Шлютер Дж., Кэннон С.Б., Абернати Б. и др. (2011)Определение сборки транскриптома и ее использование для изучения динамики генома и профилирования транскриптома у голубиного гороха ( Cajanus cajan L.). ДНК Res
18: 153–164
10.1093/dnares/dsr007
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
(2011)Определение сборки транскриптома и ее использование для изучения динамики генома и профилирования транскриптома у голубиного гороха ( Cajanus cajan L.). ДНК Res
18: 153–164
10.1093/dnares/dsr007
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
8. Гарг Р., Патель Р.К., Тьяги А.К., Джейн М. (2011) Сборка транскриптома нута De novo с использованием коротких прочтений для обнаружения генов и идентификации маркеров. ДНК Res 18: 53–63 10.1093/dnares/dsq028 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
9. Hiremath PJ, Farmer A, Cannon SB, Woodward J, Kudapa H, et al. (2011) Крупномасштабный транскриптомный анализ нута ( Cicer arietinum L.), бесхозной бобовой культуры полузасушливых тропиков Азии и Африки. Завод Биотехнолог J 9: 922–931 10.1111/j.1467-7652.2011.00625.x [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
10.
Дешам С., Ла Рота М., Раташак Дж. П., Биддл П., Турин Д. и др. (2010) Быстрое обнаружение однонуклеотидного полиморфизма по всему геному в сое и рисе путем глубокого повторного секвенирования библиотек с уменьшенным представлением с помощью анализатора генома Illumina. Ген завода
3: 53–68
10.3835/растительный геном2009.09.0026
[CrossRef] [Google Scholar]
Ген завода
3: 53–68
10.3835/растительный геном2009.09.0026
[CrossRef] [Google Scholar]
11. ван Тассел К.П., Смит Т.П.Л., Матукумалли Л.К., Тейлор Дж.Ф., Шнабель Р.Д. и соавт. (2008)Обнаружение SNP и оценка частоты аллелей путем глубокого секвенирования библиотек с уменьшенным представлением. Нат Методы 5: 247–252 10.1038/н-мет.1185 [PubMed] [CrossRef] [Google Scholar]
12. Гор М.А., Чиа Дж.М., Элшир Р.Дж., Сан К., Эрсоз Э.С. и др. (2009) Карта гаплотипов кукурузы первого поколения. Наука 326: 1115–1117 10.1126/наука.1177837 [PubMed] [CrossRef] [Академия Google]
13. Hyten DL, Cannon SB, Song Q, Weeks N, Fickus EW и др. (2010)Обнаружение SNP с высокой пропускной способностью посредством глубокого повторного секвенирования библиотеки с уменьшенным представлением для закрепления и ориентации каркасов в последовательности всего генома сои. Геномика BMC 11: 38–38 10.1186/1471-2164-11-38 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
14. Wu X, Ren C, Joshi T, Vuong T, Xu D и др. (2010)Обнаружение SNP с помощью высокопроизводительного секвенирования в сое. Геномика BMC
11: 469–469
10.1186/1471-2164-11-469[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Wu X, Ren C, Joshi T, Vuong T, Xu D и др. (2010)Обнаружение SNP с помощью высокопроизводительного секвенирования в сое. Геномика BMC
11: 469–469
10.1186/1471-2164-11-469[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
15. van Orsouw NJ, Hogers RCJ, Janssen A, Yalcin F, Snoeijers S, et al. (2007) Снижение сложности полиморфных последовательностей (CRoPS): новый подход к обнаружению крупномасштабного полиморфизма в сложных геномах. PLoS Один 2: e1172 10.1371/journal.pone.0001172 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
16. Baird NA, Etter PD, Atwood TS, Currey MC, Shiver AL, et al. (2008)Быстрое открытие SNP и генетическое картирование с использованием секвенированных маркеров RAD. PLoS Один 3: e3376 10.1371/journal.pone.0003376 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
17.
Elshire RJ, Glaubitz JC, Sun Q, Poland JA, Kawamoto K, et al. (2011) Надежный подход простого генотипирования путем секвенирования (GBS) для видов с большим разнообразием. PLoS Один
6: e19379
10.1371/journal.pone.0019379
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
PLoS Один
6: e19379
10.1371/journal.pone.0019379
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
18. Davey JW, Hohenlohe PA, Etter PD, Boone JQ, Catchen JM, et al. (2011)Полногеномное открытие генетических маркеров и генотипирование с использованием секвенирования следующего поколения. Нат Рев Жене 2: 499–510 10.1038/нрг3012 [PubMed] [CrossRef] [Академия Google]
19. Huang X, Feng Q, Qian Q, Zhao Q, Wang L и др. (2009) Высокопроизводительное генотипирование путем полногеномного повторного секвенирования. Геном Res 19: 1068–1076 10.1101/гр.089516.108 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
20. Мецкер М.Л. (2010) Технологии секвенирования – следующее поколение. Нат Рев Жене 11: 31–46 10.1038/nrg2626 [PubMed] [CrossRef] [Google Scholar]
21. Килиан Б., Гранер А. (2012)Технологии NGS для анализа разнообразия зародышевой плазмы в банках генов. Краткая функциональная геномика 11: 38–50 10.1093/bfgp/elr046 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
22. You FM, Huo N, Deal KR, Gu YQ, Luo MC и др. (2011)Обнаружение полногеномных SNP на основе аннотаций в большом и сложном геноме Aegilops tauschii с использованием секвенирования следующего поколения без эталонной последовательности генома. Геномика BMC
12: 59–59
10.1186/1471-2164-12-59
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
You FM, Huo N, Deal KR, Gu YQ, Luo MC и др. (2011)Обнаружение полногеномных SNP на основе аннотаций в большом и сложном геноме Aegilops tauschii с использованием секвенирования следующего поколения без эталонной последовательности генома. Геномика BMC
12: 59–59
10.1186/1471-2164-12-59
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
23. Гаур Р., Азам С., Джина Дж., Хан А.В., Чоудхари С. и др. (2012) Высокопроизводительное обнаружение SNP и генотипирование для построения карты насыщенного сцепления нута ( Cicer arietinum L.). ДНК Res 19: 357–373 10.1093/dnares/dss018 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
24. Хаффорд М.Б., Сюй С., ван Хеерваарден Дж., Пихаярви Т., Чиа Дж.-М. и др. (2012) Сравнительная популяционная геномика одомашнивания и улучшения кукурузы. Нат Жене 44: 808–811 10.1038/нг.2309 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
25.
Xu X, Liu X, Ge S, Jensen JD, Hu F, et al. (2012) Повторное секвенирование 50 образцов культурного и дикого риса дает маркеры для идентификации агрономически важных генов. Нат Биотехнолог
30: 105–111
10.1038/нбт.2050
[PubMed] [CrossRef] [Академия Google]
(2012) Повторное секвенирование 50 образцов культурного и дикого риса дает маркеры для идентификации агрономически важных генов. Нат Биотехнолог
30: 105–111
10.1038/нбт.2050
[PubMed] [CrossRef] [Академия Google]
26. Lam H-M, Xu X, Liu X, Chen W, Yang G и др. (2010) Повторное секвенирование генома 31 дикой и культивируемой сои выявило закономерности генетического разнообразия и селекции. Нат Жене 42: 1053–1059 10.1038/нг.715 [PubMed] [CrossRef] [Google Scholar]
27. Hiremath PJ, Kumar A, Penmetsa RV, Farmer A, Schlueter JA, et al. (2012) Крупномасштабная разработка рентабельных анализов SNP-маркеров для оценки разнообразия и генетического картирования нута и сравнительного картирования бобовых. Завод Биотехнолог J 10: 716–732 10.1111/j.1467-7652.2012.00710.x [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
28.
Ганал М.В., Дурстевиц Г., Полли А., Берар А., Баклер Э.С. и др. (2011) Большая кукуруза ( Zea mays L.) Массив генотипирования SNP: развитие и генотипирование зародышевой плазмы, а также генетические пробелы для сравнения с эталонным геномом B73. ПЛОС ОДИН
6: e28334
10.1371/journal.pone.0028334
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
ПЛОС ОДИН
6: e28334
10.1371/journal.pone.0028334
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
29. Saxena RK, Penmetsa RV, Upadhyaya HD, Kumar A, Carrasquilla-Garcia N, et al. (2009) Крупномасштабная разработка экономичных анализов маркеров однонуклеотидного полиморфизма для генетического картирования голубиного гороха и сравнительного картирования бобовых. ДНК Res 19: 449–461 10.1093/dnares/dss025 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
30. Yu H, Xie W, Li J, Zhou F, Zhang Q (2014) Полногеномный массив SNP (RICE6K) для геномной селекции риса. Завод Биотехнолог J 12: 28–37 10.1111/пби.12113 [PubMed] [CrossRef] [Google Scholar]
31. Ахунов Э., Николет С., Дворжак Дж. (2009) Генотипирование однонуклеотидного полиморфизма у полиплоидной пшеницы с помощью анализа Illumina GoldenGate. Теория Appl Genet 119: 507–517 10.1007/s00122-009-1059-5 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
32.
Гупта П. К., Рустги С., Мир Р.Р. (2008) Высокопроизводительные ДНК-маркеры на основе массива для улучшения урожая. Наследственность
101: 5–18
10.1038/hdy.2008.35
[PubMed] [CrossRef] [Google Scholar]
К., Рустги С., Мир Р.Р. (2008) Высокопроизводительные ДНК-маркеры на основе массива для улучшения урожая. Наследственность
101: 5–18
10.1038/hdy.2008.35
[PubMed] [CrossRef] [Google Scholar]
33. Ганал М.В., Полли А., Гранер Э.М., Плиске Дж., Визеке Р. и др. (2012)Большие массивы SNP для генотипирования сельскохозяйственных растений. Джей Биоски 37: 821–828 10.1007/с12038-012-9225-3 [PubMed] [CrossRef] [Google Scholar]
34. Бланка Дж. М., Паскуаль Л., Зиарсоло П., Нуэз Ф., Каньисарес Дж. (2011) Магистраль ngs: конвейер для очистки чтения, сопоставления и вызова snp с использованием последовательности следующего поколения. Геномика BMC 12: 285 10.1186/1471-2164-12-285 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
35. Deng X (2011) Seqgene: комплексное программное решение для анализа данных секвенирования экзома и транскриптома. БМК Биоинформатика 12: 267 10.1186/1471-2105-12-267 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
36. Сана М.Э., Ясконе М., Марчетти Д., Палатини Дж., Галассо М. и др. (2011) Games идентифицирует и аннотирует мутации в проектах секвенирования следующего поколения. Биоинформатика
27: 9–13
10.1093/биоинформатика/btq603
[PubMed] [CrossRef] [Академия Google]
Сана М.Э., Ясконе М., Марчетти Д., Палатини Дж., Галассо М. и др. (2011) Games идентифицирует и аннотирует мутации в проектах секвенирования следующего поколения. Биоинформатика
27: 9–13
10.1093/биоинформатика/btq603
[PubMed] [CrossRef] [Академия Google]
37. Qi J, Zhao F, Buboltz A, Schuster SC (2010) ingap: интегрированный конвейер анализа генома следующего поколения. Биоинформатика 26: 127–129 10.1093/биоинформатика/btp615 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
38. ДеПристо М.А., Бэнкс Э., Поплин Р., Гаримелла К.В., Магуайр Дж.Р. и др. (2011)Структура для обнаружения вариаций и генотипирования с использованием данных секвенирования ДНК следующего поколения. Нат Жене 43: 491–498 10.1038/нг.806 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
39.
Cock PJA, Fields CJ, Goto N, Heuer ML, Rice PM (2010) Формат файла Sanger FASTQ для последовательностей с показателями качества и варианты Solexa/Illumina FASTQ. Нуклеиновые Кислоты Res
38: 1767–1771 гг. 10.1093/нар/гкп1137
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
10.1093/нар/гкп1137
[Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
40. Формат Fastq. Доступно: http://en.wikipedia.org/wiki/FASTQ_format/. По состоянию на октябрь 2013 г.
41. Ли Х, Дурбин Р. (2010) Быстрое и точное выравнивание с длинным чтением с преобразованием Берроуза-Уилера. Биоинформатика 26: 589–595 10.1093/биоинформатика/btp698 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
42. Li H, Ruan J, Durbin R (2008) Картирование коротких чтений секвенирования ДНК и вызов вариантов с использованием показателей качества картирования. Геном Res 18: 1851–1858 гг. 10.1101/гр.078212.108 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
43. Лангмид Б., Зальцберг С.Л. (2012) Быстрое выравнивание с промежутками чтения с Bowtie 2. Nat Methods 9: 357–359 10.1038/нмет.1923 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
44. Короткое выравнивание Novocraft. Доступно: http://www.novocraft.com/. По состоянию на октябрь 2013 г.
45. Li R, Yu C, Li Y, Lam T-W, Yiu S-M (2009) SOAP2: улучшенный сверхбыстрый инструмент для выравнивания коротких чтений. Биоинформатика 25: 1966–1967 10.1093/биоинформатика/btp336 [PubMed] [CrossRef] [Google Scholar]
46. SOAP : Пакет для краткого анализа олигонуклеотидов. Доступно: http://soap.genomics.org.cn/soapaligner.html. По состоянию на октябрь 2013 г.
47. Ли Х., Хэндсакер Б., Высокер А., Феннелл Т., Руан Дж. и др. (2009 г.) Формат выравнивания/карты последовательности и SAMtools. Биоинформатика 25: 2078–2079 гг. 10.1093/биоинформатика/btp352 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
48.
Азам С., Такур В., Руперао П., Шах Т., Баладжи Дж. и др. (2012) Консенсусный вызов на основе охвата (CbCC) чтения коротких последовательностей и сравнение результатов CbCC для идентификации SNP в нуте ( Cicer arietinum ; Fabaceae ), культурных видах без эталонного генома. Ам Джей Бот
99: 186–192
10.3732/ajb. 1100419[PubMed] [CrossRef] [Google Scholar]
1100419[PubMed] [CrossRef] [Google Scholar]
49. Геномы. Доступно: http://www.1000genomes.org/node/101. По состоянию на октябрь 2013 г.
50. Li R, Li Y, Fang X, Yang H, Wang J и др. (2009)Обнаружение SNP для массивно-параллельного ресеквенирования всего генома. Геном Res 19: 1124–1132 10.1101/гр.088013.108 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
51. Милн И., Байер М., Кардл Л., Шоу П., Стивен Г. (2010) Планшет — визуализация сборки последовательности нового поколения. Биоинформатика 26: 401–402 10.1093/биоинформатика/btp666 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
52. Милн И., Шоу П., Стивен Г., Байер М., Кардл Л. и др. (2010) Flapjack – графическая визуализация генотипа. Биоинформатика 26: 3133–3134 10.1093/биоинформатика/btq580 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
53. Версия стандартного формата функций 3 (GFF3). Доступно: http://www.sequenceontology.org/gff3.shtml. По состоянию на октябрь 2013 г.
54. Нильсен Р., Пол Дж. С., Альбрехтсен А., Сонг Ю. С. (2011) Генотип и вызов SNP на основе данных секвенирования следующего поколения. Нат Рев Жене 12: 443–451 10.1038/нрг2986 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
55. Инструмент для разработки ПЦР (ADT). Доступно: http://support.illumina.com/array/array_software/assay_design_tool.ilmn. По состоянию на октябрь 2014 г.
56. Фличек П., Бирни Э. (2009) Смысл чтения последовательности: методы выравнивания и сборки. Нат Методы 6: 6–6 10.1038/н-мет.1376 [PubMed] [CrossRef] [Google Scholar]
57. Li H, Homer N (2010) Обзор алгоритмов выравнивания последовательностей для секвенирования следующего поколения. Кратко Биоинформ 11: 473–483 10.1093/нагрудник/барбекю015 [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
58.
Брэдбери П.Дж., Чжан З., Крун Д.Э., Касстевенс Т.М., Рамдосс Ю. и др. (2007) TASSEL: программное обеспечение для картирования ассоциаций сложных признаков в различных образцах.